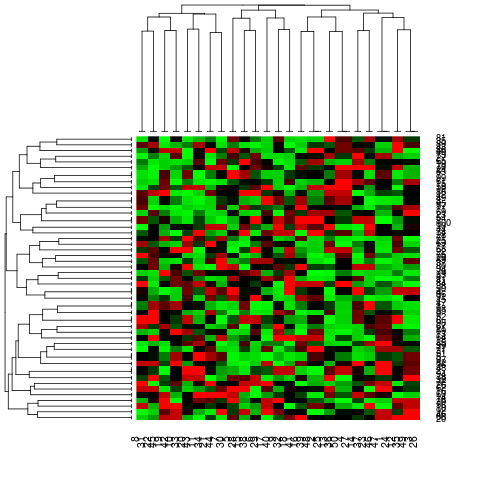
DNAマイクロアレイのプロットの課題をやってみた。gplotsライブラリのheatmap.2関数を使う場合と点をたくさん描くゴリ押しバージョンの2つを試した。
#-------------------- # 発現強度(0から1の範囲) #-------------------- library("dplyr") sig.intensity <- rnorm(n=5000, mean=10, sd=1) %>% matrix(nr=100, nc=50) sig.intensity <- (sig.intensity - min(sig.intensity)) sig.intensity <- sig.intensity / max(sig.intensity) # 発現強度matrixにラベルをつける rname <- c() for (i in 1:100) { rname <- c(rname, paste("gene", as.character(i))) } cname <- c() for (i in 1:50) { cname <- c(cname, paste("sample", as.character(i))) } dimnames(sig.intensity) <- list(rname, cname) #--------------- # 緑、赤の混合比 #--------------- # ランダムな場合 sig.color <- runif(n=5000, min=0, max=1) %>% matrix(nr=100, nc=50) # 遺伝子で相関がある場合 n.cluster <- runif(n=1, min=30, max=70) cluster <- sample(1:n.cluster, prob=rep(1/n.cluster, n.cluster), replace=T) cluster.color <- runif(n=n.cluster, min=0, max=1) sig.color <- cluster.color[cluster] %>% jitter %>% matrix(nr=100, nc=50) #--------------- # プロット #--------------- # heatmap(発現強度は無視) library("gplots") heatmap.2(sig.color, col=redgreen(256), trace="none") # greenred(100)? # プロット(ゴリ押し) y <- c(); for (i in 100:1) { y <- c(y, rep(i, 50)) } plot(x = rep(1:50, 100), y = y, pch = 16, col = rgb(sig.color*sig.intensity, 1-sig.color*sig.intensity, 0), cex=1) #あまりうまくいっていない