RのsurvivalパッケージにあるcolonはStage B/Cの結腸癌患者を対象とした術後補助化学療法の比較臨床試験データ。pythonを使ってこのデータの生存時間解析をやってみる。まずは可視化から。
colon.csvの様子
id,study,rx,sex,age,obstruct,perfor,adhere,nodes,status,differ,extent,surg,node4,time,etype 1,1,Lev+5FU,1,43,0,0,0,5,1,2,3,0,1,1521,2 1,1,Lev+5FU,1,43,0,0,0,5,1,2,3,0,1,968,1 2,1,Lev+5FU,1,63,0,0,0,1,0,2,3,0,0,3087,2 2,1,Lev+5FU,1,63,0,0,0,1,0,2,3,0,0,3087,1 3,1,Obs,0,71,0,0,1,7,1,2,2,0,1,963,2 3,1,Obs,0,71,0,0,1,7,1,2,2,0,1,542,1 4,1,Lev+5FU,0,66,1,0,0,6,1,2,3,1,1,293,2 4,1,Lev+5FU,0,66,1,0,0,6,1,2,3,1,1,245,1 5,1,Obs,1,69,0,0,0,22,1,2,3,1,1,659,2
とりあえずそれぞれプロットしてみる。データ処理はpandasでゴリ押ししたかったが触り始めたばかりなので最初の方だけ。
from __future__ import division import numpy as np import pandas as pd import matplotlib.pyplot as plt #read data colon = pd.read_csv('colon.txt') mask = np.array([i == 2 for i in colon.etype]) cd = colon[mask][['rx','time']] lev = np.array(cd[[i == 'Lev' for i in cd.rx]].time) lev5fu = np.array(cd[[i == 'Lev+5FU' for i in cd.rx]].time) obs = np.array(cd[[i == 'Obs' for i in cd.rx]].time) #plot def survival_function(arr): return [1 - i/len(arr) for i in range(len(arr))] fig = plt.figure() ax = fig.add_subplot(111) ax.set_xlabel('days') ax.set_ylabel('survival_function') for arr, name in zip((lev, lev5fu, obs),('Lev', 'Lev+5FU', 'Obs')): ax.plot(np.sort(arr), survival_function(arr), label=name) ax.legend(['Lev', 'Lev+5FU', 'Obs']) plt.show()
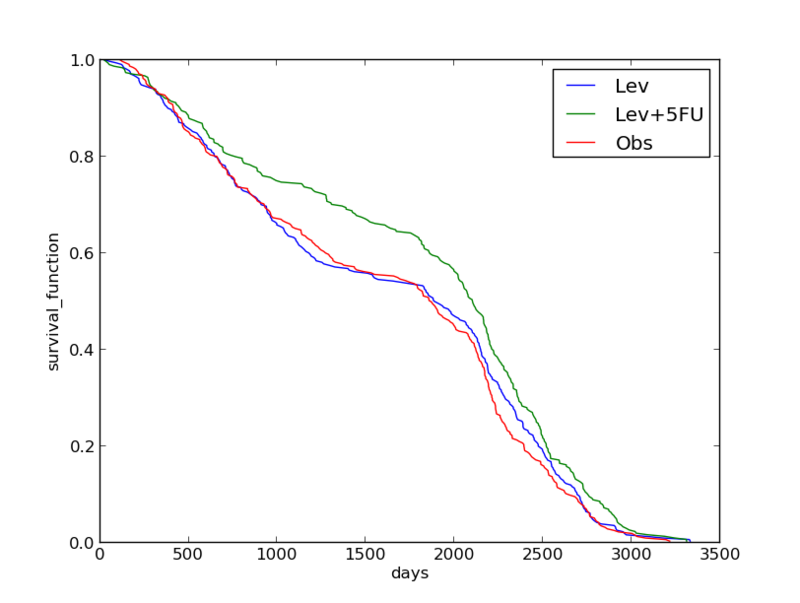
Levamisole + 5-FUの結果が良さそう。
PMF(probability mass function: 確率質量関数)とCDF(cumulative distribution function:累積分布関数)
確率質量関数(pmf)は正規化されたヒストグラムのことを指す。今回はまず死亡するまでの日数のヒストグラムを考え、さらにこれを正規化することにする。累積分布関数(cdf)はpmfをパーセンタイル順位に対応させたもの。
- numpy.histogramを使った場合
テキストにはnp.histogramの引数にnormedを使っているがこれはnumpy2.0で廃止予定の機能らしいのでdensityを使う。densityだと面積の和が1になるように密度を返してくる。今回はそれぞれのビンごとの和が1になればいいので\(\rm \overline{pdf}\)を求めてからpmfにビンの幅を掛けて変換する。cdfはpmfのcumsumで求める。
#plot pmf fig = plt.figure() ax_hist = fig.add_subplot(311) ax_pmf = fig.add_subplot(312) ax_cdf = fig.add_subplot(313) ax_hist.set_ylabel('hist') ax_pmf.set_ylabel('pmf') ax_pmf.tick_params(labelbottom='off') ax_cdf.set_ylabel('cdf') ax_cdf.set_xlabel('days') for i, arr in enumerate([lev, lev5fu, obs]): #histogram hist, bin_edges = np.histogram(arr, bins=35, range=(0,3500)) #pmf, cdf pdf, bin_edges = np.histogram(arr, bins=35, range=(0,3500), density=True) pmf = pdf*100 cdf = np.cumsum(pmf) #define width of bars(== range[1]/bins/3) width = bin_edges[-1]/105 ax_hist.bar(bin_edges[:-1]+i*width, hist, width=width, color=str(i/4)) ax_hist.legend(['Lev', 'Lev+5FU', 'Obs']) ax_pmf.bar(bin_edges[:-1]+i*width, pmf, width=width, color=str(i/4)) ax_cdf.bar(bin_edges[:-1]+i*width, cdf, width=width, color=str(i/4)) plt.show()
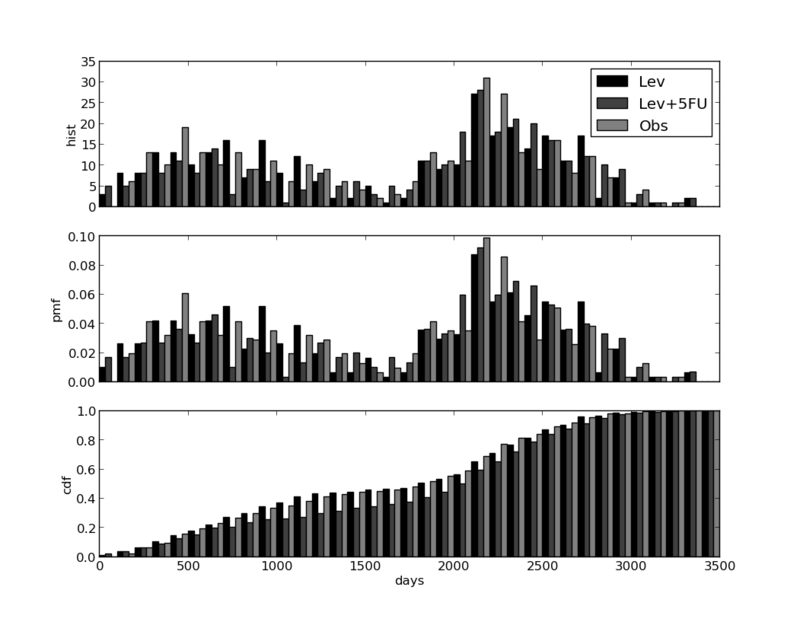
これは分かりにくい。pmfは二峰性らしい。
- matplotlib.pyplot.histを使う場合
np.histogramの場合と同様、pmfを求める際にはnormed=Trueではなく、weightsを設定する必要がある。
for i, arr in enumerate([lev, lev5fu, obs]): #histogram hist, bin_edges = np.histogram(arr, bins=35, range=(0,3500)) #define width of bars(== range[1]/bins/3) width = bin_edges[-1]/105 ax_hist.bar(bin_edges[:-1]+i*width, hist, width=width, color=str(i/4)) ax_hist.legend(['Lev', 'Lev+5FU', 'Obs']) #pmf ax_pmf.hist(arr, bins=50, range=(0,3500), weights=np.array([1/len(arr)]*len(arr)), histtype='step', color=('b','g','r')[i]) ax_pmf.legend(['Lev', 'Lev+5FU', 'Obs']) #cdf ax_cdf.hist(arr, bins=100, range=(0,3500), normed=True, cumulative=True, histtype='step', color=('b','g','r')[i]) plt.show()
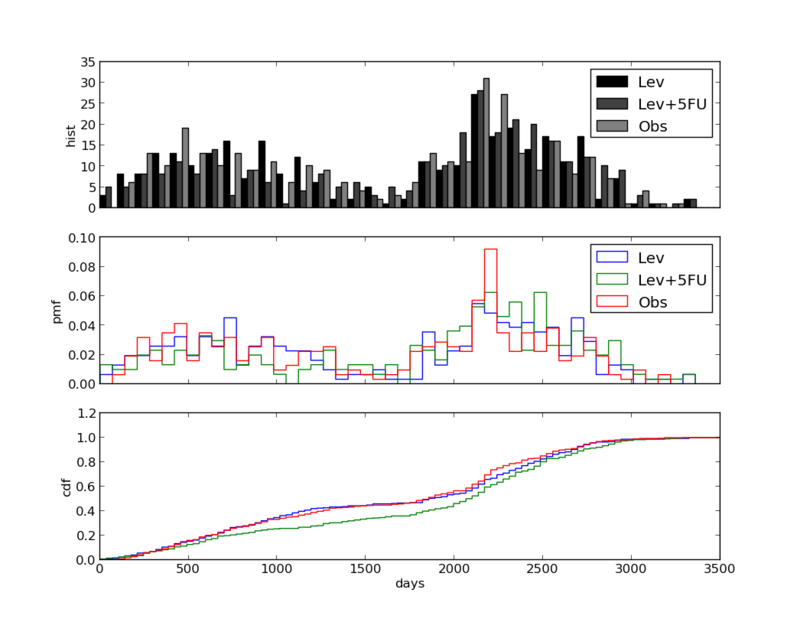
そもそもpmfは今回のような標本数の多いケースでは可視化に向いていないことが分かる。